Sie sind hier: Startseite > Diagnostik > Urin-Untersuchung > Urinkultur
Urinkultur: Keimzahl, Identifizierung der Bakterien und Antibiogramm
- Urinuntersuchung: Urinsediment
- Urinuntersuchung: Urinkultur
- Urinuntersuchung: 24 h-Urin
- Urinuntersuchung: Urinzytologie
Indikationen für eine Urinkultur
Bei asymptomatischen Patienten:
- Leukozyturie, Hämaturie oder positiver Nitrittest bei Patienten mit Risikofaktoren (Immunsuppression, vesikoureteraler Reflux, Harnblasenentleerungsstörungen)
- Schwangerschaft (Erstvorstellung und 16. SSW)
- Vor und nach interventionellen Eingriffen an den Harnwegen
- Nach Antibiotikatherapie bei Schwangeren, Männern, Pyelonephritis oder komplizierter Harnwegsinfektion
Bei symptomatischen Patienten:
- Alle Patienten mit Verdacht auf Harnwegsinfektion, bei Frauen mit unkomplizierter Zystitis kann auf die Kultur verzichtet werden.
- Bei unklaren Abdominalbeschwerden oder Flankenschmerz
- Bei rezidivierenden Harnwegsinfektion
- Bei Zeichen einer nosokomialen Harnwegsinfektion
- Fehlender Therapieerfolg, Fieber oder Sepsis
Uringewinnung:
Das Risiko einer bakteriellen Kontamination beträgt bei nichtinvasiver Uringewinnung bis zu 40%, je nach Geschlecht, Alter und Compliance. Mittel der ersten Wahl für die Uringewinnung ist die sterile Uringewinnung im Rahmen von Katheterisierungen (Einmalkatheterurin), Interventionen oder durch eine suprapubische Blasenpunktion. Ansonsten wird der Urin mittels Mittelstrahlurin und bei Säuglingen mittels Klebebeutel gewonnen.
4 Gläser-Probe:
Bei V. a. chronische Prostatitis werden 4 Portionen einer Miktion getrennt gesammelt und mikrobiologisch untersucht (Sedimentmikroskopie und Kultur).
- Die ersten 10 ml Urin (Harnröhrenkeime)
- Mittelstrahlurin (Harnblasenkeime)
- Prostataexprimat (Prostatakeime): Normalerweise unter 10 Leukozyten pro Gesichtsfeld in hoher Vergrößerung.
- Die ersten 10 ml Urin nach Prostatamassage (Prostatakeime)
Aufgrund des hohen Aufwandes und der Kosten hat sich der Zwei-Gläser-Test durchgesetzt:
- Mittelstrahlurin (Harnblasenkeime)
- Die ersten 10 ml Urin nach der Prostatamassage (Prostatakeime)
Probentransport:
Die Verarbeitung von Nativurin innerhalb von 2 h ist optimal, bei längeren Transportzeiten muss der Urin gekühlt werden oder in speziellen Monovetten (Stabilisator Borsäure) gelagert werden.
Quantitative Urinkultur
 |
Die Keimzahl wird mit Hilfe einer quantitativen Urinkultur bestimmt. Die am häufigsten verwendete Technik benutzt spezielle kalibrierte Ösen, mit deren Hilfe werden 10 μl Urin auf einer Agarplatte (z.B. CLED-Agar) ausgestrichen [Abb. quantitative Urinkultur]. Die Platten werden 24 h bei 36 Grad Celsius bebrütet. Eine Kolonie entspricht 102 KBE/ml, 10 Kolonien 103 KBE/ml, 100 Kolonien 104 KBE/ml u. 1000 Kolonien 105 KBE/ml. Die Keimzahl wird semiquantitativ durch Vergleich mit standardisierten Abbildungen bestimmt. Die Keimzahlbestimmung mit Hilfe von fertigen Nährbodenträgern (Urintauchkultur wie z.B. Urikult) ist von geringer Genauigkeit und sollte die Ausnahme bleiben.
CLED-Agar:
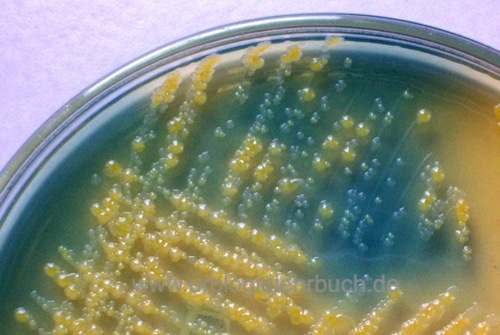
Dieser unselektive Agar wird häufig für die quantitative Urinkultur verwendet, er ermöglicht das Wachstum von allen typischen Bakterien für eine Harnwegsinfektion. CLED steht für Cystine Lactose Electrolyte Deficient Agar, der niedrige Elektrolytgehalt verhindert das Schwärmen von Proteus. Laktosefermentierende Bakterien bilden gelbe Kolonien und färben den Agar gelb, Nonfermenter bilden blaugraue Kolonien und der Agar bleibt bläulich gefärbt [Abb. Mischkultur].
Kass-Zahl:
105 Bakterien pro ml in einem sauber gewonnenen Mittelstrahlurin sind ein Zeichen für eine klinisch signifikante Harnwegsinfektion. Bei hoher Urinausscheidung oder steriler Uringewinnung können auch kleinere Bakterienzahlen ein signifikanter Hinweis auf eine Harnwegsinfektion sein.
Identifizierung der Bakterien in einer Urinkultur
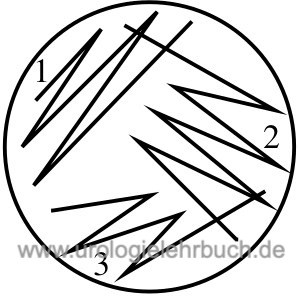
Eine Identifizierung der Bakterien ist nur dann sinnvoll, wenn kein gemischtes Bakterienwachstum (mehr als zwei Keime) vorliegt. Gemischtes Bakterienwachstum spricht für eine Kontamination während der Uringewinnung und die Identifizierung wird nicht durchgeführt. Die Urinkultur sollte dann neu abgenommen werden. Die korrekte Identifizierung der Bakterien gelingt nur dann sicher, wenn die Kolonien auf u.g. Agarplatten vereinzelt wachsen. In konfluenten Kolonien können verschiedene Keime unbemerkt gemischt wachsen. Im Zweifelsfall muss nochmals ein fraktionierter 3-Ösen-Ausstrich zur Vereinzelung der Kolonien durchgeführt werden [Abb. 3-Ösen-Ausstrich].
Idealerweise werden bereits bei der quantitativen Urinkultur zusätzlich folgende Selektivnährböden beimpft:
MacConkey-Agar:
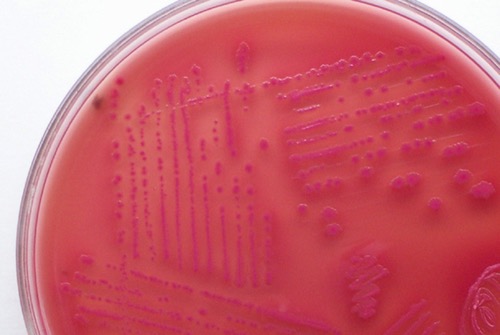
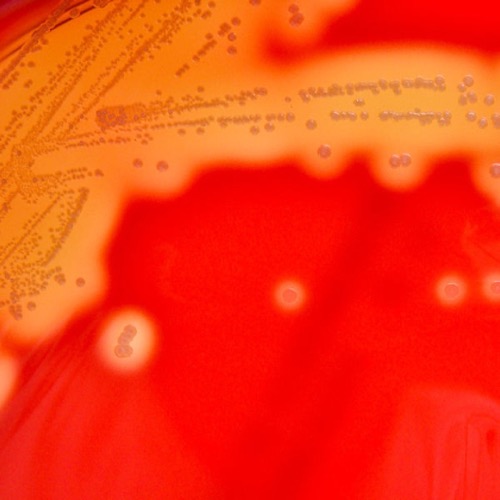
Der MacConkey-Agar ist ein Selektivnährböden für das bevorzugte Wachstum gramnegativer Bakterien. Lakosefermentierende Bakterien färben sich durch den enthaltenen Indikator rot-violett [Abb. MacConkey-Agar], Nonfermenter bilden graue Kolonien.
 |
Columbia-Blut-CNA-Agar:
Der Columbia-Blut-CNA-Agar ist ein Selektivnährmedium für das bevorzugte Wachstum grampositiver Bakterien (v.a. Streptokokken, Enterokokken und Staphylokokken). Der Agar enthält intakte Erythrozyten und die Antibiotika Colistin (Polymyxin E) und Nalidixinsäure, die das Wachstum gramnegativer Bakterien hemmen. Die Bakterien können weiter nach ihrem Hämolyseverhalten eingegrenzt werden: α-Hämolyse (Vergrünung durch Oxidation des Hämoglobins), β-Hämolyse (Aufhellung durch Lyse der Erythrozyten) und γ-Hämolyse (unveränderte Agardichte) [Abb. β-Hämolyse im Columbia-Blut-CNA-Agar].
 |
Sabouraud-Dextrose-Agar:
Sabouraud-Dextrose-Agar ist ein Selektivnährmedium, auf dem bevorzugt Hefen und Schimmelpilze wachsen.
Indentifizierung der Bakterienart:
Erste Hinweise auf die Bakterienart einer Urinkultur ergeben sich durch das Wachstumsmuster auf den oben genannten Agarplatten und mit Hilfe einfacher chemischer Testreaktionen:
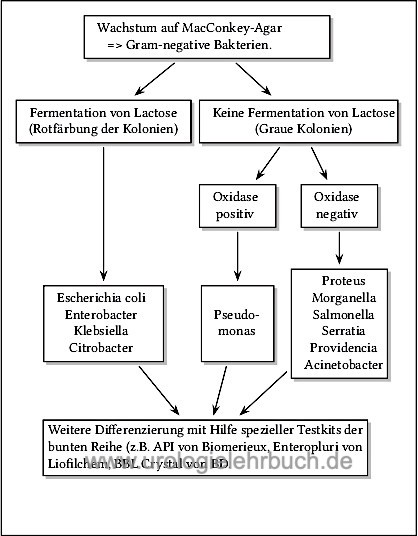
- Wachstum auf MacConkey-Agar → gramnegative Stäbchen. Eine weitere Differenzierung ist mittels Lactose-Reaktion und der Oxidase-Reaktion möglich [Abb. Indentifizierung von gramnegativen Bakterien].
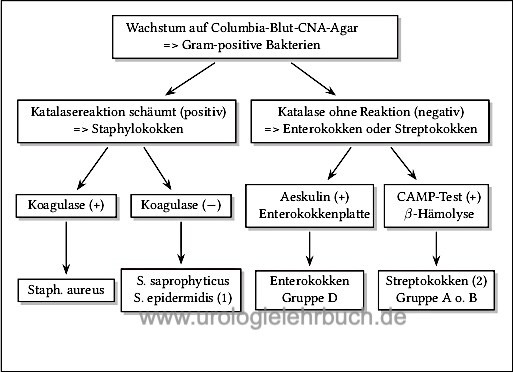
- Wachstum auf Columbia-Blut-CNA-Agar → grampositive Kokken. Eine weitere Differenzierung ist mittels Katalase-Reaktion, der Hämolysereaktion, der Aeskulinspaltung, des CAMP-Tests und der Novobiocin- oder Bacitracin-Empfindlichkeit möglich [Abb. Indentifizierung von grampositiven Bakterien].
- Wachstum auf Sabouraud-Dextrose-Agar: in der Regel Hefepilze wie Candida, weitere Differenzierung und Resistenzprüfung mit Hilfe von Testkits.
 |
Bakterienidentifizierung mit der "Bunten Reihe"
Bakterien können mit Hilfe spezieller Testkits (sogenannte bunte Reihe) weiter differenziert werden. Je nach biochemischen Eigenschaften (Oxidase positiv/negativ, Laktosefermentation, grampositiv) werden unterschiedliche Testkits verwendet. Eine definierte Menge von Einzelkolonien einer Reinkultur wird in NaCl resuspendiert. Die Bakterienmenge wird anhand der Trübung (McFarland Standard 0,5) oder genauer mit der Photometrie bestimmt. Die Bakterienlösung wird in das Testkit pipettiert und und über Nacht im Brutschrank inkubiert. Gleichzeitig wird eine Kontrollplatte mit der Bakteriensuspension beimpft, um die Reinheit des Inokulums zu überprüfen. Die bunte Reihe nutzt die unterschiedlichen biochemischen Leistungen der Bakterien, diese werden mit Hilfe von Farbreaktionen für den Untersucher sichtbar. Untersucht werden unter anderem Enzymaktivitäten (Oxidase, Katalase, Laktase, Gärung, Urease), Mobilität oder die Verwendung von Citrat als Energielieferant. Ein häufig verwendetes System ist API (Analytic Profile Index) oder das VITEK-System, das von der Firma bioMérieux vertrieben wird. Eine Alternative ist das RAS-ID System von der Firma BIO-RAD.
Interne Qualitätssicherung:
Die Methodik der Bakterienidentifizierung muss mit Hilfe von definierten Referenzkulturen (Kontrollstämme) bei Chargenwechsel überprüft werden. Die Referenzkulturen sind in kryokonservierter Form käuflich erhältlich, mindestens monatlich müssen frische Stammkulturen daraus hergestellt werden. Die daraus gezogene Gebrauchskultur darf längstens eine Woche verwendet werden. Bei jeder Identifizierung muss zwingend die Inokulumsreinheit durch eine Kontrollplatte überprüft werden. Es ist eine Statistik über die Häufigkeit der nachgewiesenen Erreger zu führen (RiliBÄK 2014).
Externe Qualitätssicherung:
Halbjährliche Teilnahme an Ringversuchen.
Prüfung auf antibakterielle Hemmstoffe
Für die Prüfung auf antibakterielle Hemmstoffe werden Agarplatten verwendet, welche Sporen von Bacillus subtilis enthalten. Filterplättchen mit 10 μl Urin werden auf die Agarplatten gelegt und 24 h bei 36 Grad Celsius bebrütet, die Ausbildung von Hemmhöfen weist auf antibakterielle Hemmstoffe hin.
Resistenzbestimmung oder Antibiogramm
Eine definierte Menge von einzelnen Kolonien einer Reinkultur wird in NaCl resuspendiert. Die Bakterienmenge wird anhand der Trübung abgeschätzt (McFarland Standard 0,5) oder genauer mit der Photometrie bestimmt. Die Bakteriensuspension wird mit Hilfe eines Wattetupfers auf einer Müller-Hinton Agarplatte lückenlos ausgestrichen. Ziel ist ein gleichmäßiges und konfluentes Wachstum der Bakterienkolonien. Zusätzlich wird von der Bakteriensuspension eine Kontrollplatte beimpft, um die Inokulumsreinheit zu überprüfen. Filterplättchen mit den gängigen Antibiotika (Agardiffusionstest) werden auf die Müller-Hinton Agarplatte gelegt und die Platten werden 24 h bei 36 Grad Celsius bebrütet. Das Antibiotikum diffundiert aus dem Filterplättchen in den Agar und bildet in Abhängigkeit der Empfindlichkeit einen unterschiedlich großen Hemmhof.
EUCAST:
Die Resistenztestung ist in Europa durch EUCAST (European Committee on Antimicrobial Susceptibility Testing) standardisiert. Auf der Internetseite https://www.eucast.org werden detailliert die Technik der Resistenztestung und die Durchmesser der Hemmhöfe für die Auswertung der Sensibilität und Resistenz publiziert und jährlich aktualisiert. Für die Resistenzbestimmung gibt es auch mehrere kommerzielle Systeme, die das Verfahren zu vereinfachen (z.B. RAS-ID System von der Firma BIO-RAD oder VITEK-System von bioMérieux).
Interne Qualitätssicherung:
Die Methodik der Resistenztestung muss mit Hilfe von definierten Referenzkulturen (Kontrollstämme) wöchentlich und bei Chargenwechsel überprüft werden. Die Referenzkulturen sind in kryokonservierter Form käuflich erhältlich, mindestens monatlich müssen frische Stammkulturen daraus hergestellt werden. Die daraus gezogene Gebrauchskultur darf längstens eine Woche verwendet werden. Es ist eine Statistik über die Empfindlichkeit der nachgewiesenen Erreger gegenüber Antibiotika zu führen (RiliBÄK 2014).
Externe Qualitätssicherung:
Halbjährliche Teilnahme an Ringversuchen.
| Urinsediment | Suchen | 24 h-Sammelurin |
Sachregistersuche: A B C D E F G H I J K L M N O P Q R S T U V W X Y Z
Literatur Urinkultur
 English Version: Urine culture and 24-hour urine
English Version: Urine culture and 24-hour urine
Urologielehrbuch.de ohne Werbung
Diese Internetseite ermöglicht mit Hilfe von Werbung den Volltext-Zugriff auf das aktuelle Urologielehrbuch.de. Viele Bilder sind zum Schutz von Laien verpixelt oder ausgeblendet. Regelmäßig wiederkehrende (fachkundige) Leser können die Werbebanner abschalten und Zugriff auf alle Abbildungen erhalten: Werden Sie Mitglied über die Crowdfunding-Plattform Steady und unterstützen Sie damit Urologielehrbuch.de.
Urologielehrbuch.de als Hardcover-Buch
 Aktuell, detailliert und übersichtlich: Urologielehrbuch.de wird auch als hochwertiges Hardcover-Buch veröffentlicht. Die 17. Auflage (Ausgabe 2024) ist seit Oktober 2024 verfügbar, siehe Abschnitt Neuigkeiten für die Aktualisierungen und Links für den Buchkauf.
Aktuell, detailliert und übersichtlich: Urologielehrbuch.de wird auch als hochwertiges Hardcover-Buch veröffentlicht. Die 17. Auflage (Ausgabe 2024) ist seit Oktober 2024 verfügbar, siehe Abschnitt Neuigkeiten für die Aktualisierungen und Links für den Buchkauf.